L’importance d’un nom
Publié
Catégories
Auteur
Combien d'entre nous, en voyant un oiseau
Combien d’entre nous, en observant un oiseau, une plante ou un insecte, prennent le temps de s’interroger : De quoi s’agit-il ? Quelle est cette espèce ? Comment la classer ? La relation entre l’humanité et la nature est essentielle. Notre besoin de nommer et de catégoriser nos voisins non humains reflète une curiosité profonde, bien que les appellations locales varient largement selon les régions et les cultures. En réalité, la manière dont nous répondons à ces questions révèle beaucoup sur notre environnement et les communautés avec lesquelles nous vivons.
Le poisson d’eau douce communément appelé spare tête-de-mouton en Ontario est connu sous le nom de Gaspergoo en Louisiane. À Toronto, tout petit poisson argenté peut être désigné comme un vairon, bien que les passionnés et les scientifiques ne distinguent pas moins de 38 espèces différentes de vairons dans la seule province de l’Ontario — du mulet perlé à la queue à tache noire. Pour aller encore plus encore loin, les cultures Tukano du nord-ouest de l’Amazonie sont réputées pour la finesse avec laquelle elles identifient chacune des centaines d’espèces de poissons tropicaux qui peuplent les ruisseaux et les rivières de leur forêt. Cette précision fait écho à celle des cultures inuites du Canada, capables de reconnaître les multiples types de neige et de glace qui façonnent leur environnement.
Les nombreuses façons dont les humains nomment les éléments qui constituent la nature reflètent la richesse et la diversité des cultures et des points de vue. Toutefois, pour les spécialistes de la biodiversité qui doivent recueillir, organiser et transmettre des données sur les espèces de manière précise et efficace à travers les générations, les cultures et les communautés, il est essentiel de disposer d’un système complémentaire permettant une nomenclature cohérente et rigoureuse. De nouveaux outils sont également nécessaires pour identifier rapidement et correctement chaque espèce.
La perte accélérée de biodiversité à l’échelle mondiale place tragiquement les scientifiques dans une course contre la montre : il ne s’agit plus seulement de protéger les espèces connues, mais aussi de découvrir, différencier et nommer l’incroyable diversité encore non identifiée. Plus que jamais, les biologistes ont besoin du soutien des technologies émergentes et des mégadonnées pour attribuer rapidement et avec précision un nom scientifique à la multitude d’espèces qui peuplent la Terre. Les conservateurs et les collections d’histoire naturelle du ROM, ainsi que les recherches de pointe qu’ils mènent et facilitent, jouent un rôle essentiel dans cette mission et contribuent à ouvrir la voie vers une meilleure compréhension et protection du vivant.
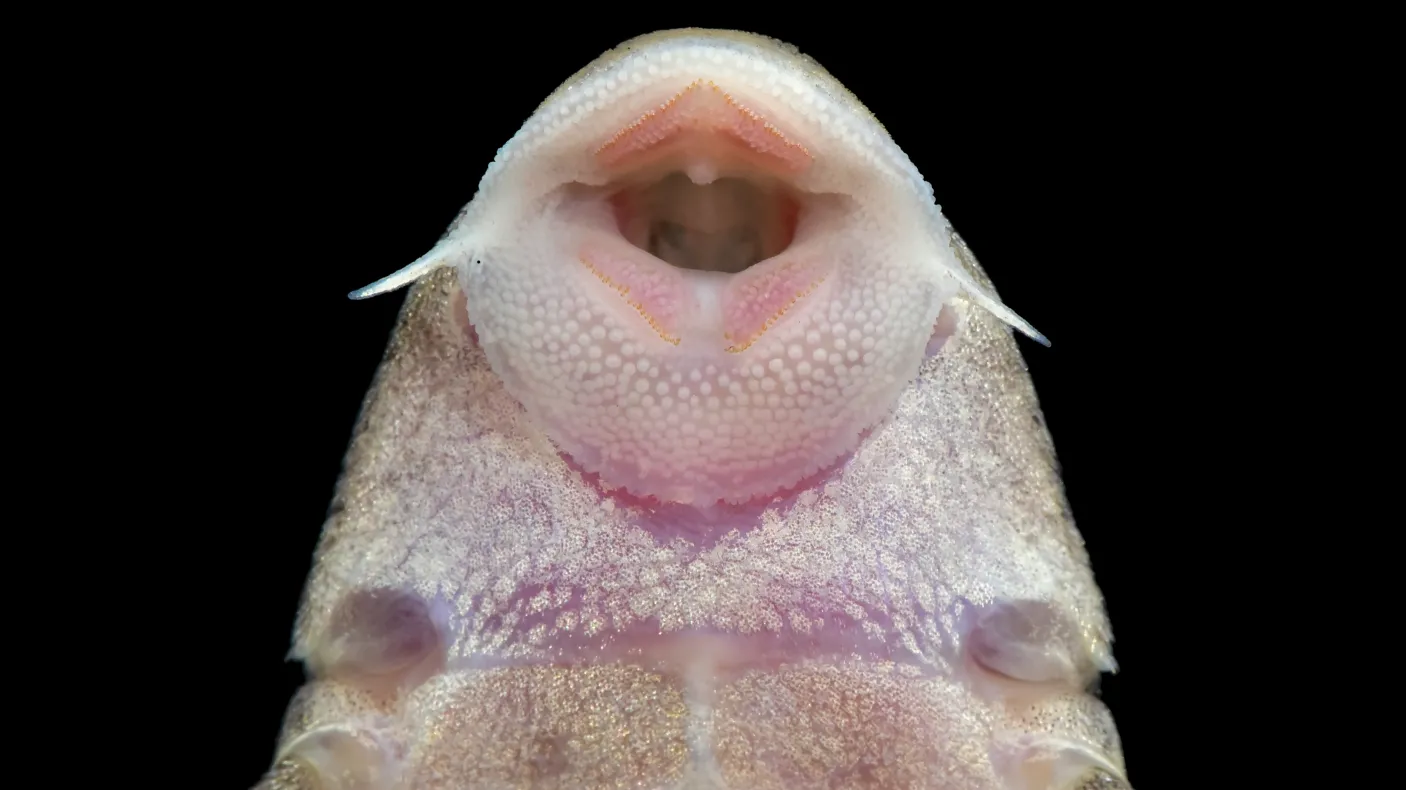
Une grande partie de mes recherches récentes, en tant que conservateur associé des poissons au ROM, porte sur la clarification des identifications taxonomiques et l’amélioration des outils de reconnaissance des espèces de poissons en Amazonie occidentale, notamment dans l’est de l’Équateur et le nord du Pérou. Les rivières de cette région comptent parmi les plus riches en biodiversité du bassin amazonien — lui-même l’écosystème d’eau douce le plus diversifié de la planète. Alors que les biologistes de la pêche en Ontario peinent parfois à distinguer quelques dizaines d’espèces de vairons — une infime portion des quelque 5 000 espèces de poissons recensées en Amazonie — les scientifiques et les acteurs de la conservation, tant locaux qu’internationaux, doivent désormais s’appuyer sur des technologies innovantes pour identifier et surveiller rapidement, mais avec précision, les populations de poissons menacées par les barrages, l’exploitation minière, la déforestation et la sécheresse.
Travailler avec l’ADN environnemental, c’est un peu comme manipuler de la magie. On a l’impression d’avoir été propulsés dans un univers futuriste digne de Star Trek, où tout organisme ou objet peut être identifié instantanément grâce à un simple balayage de tricordeur.
ADN environnemental
Le métabarcoding (ou identification par signature ADN) de l’ADN environnemental (ADNe), est une méthode de pointe que nous développons pour répondre à ce besoin, en collaboration avec les étudiant.e.s et le personnel de la division des poissons du ROM, ainsi que nos partenaires en Équateur et au Pérou. Cette approche novatrice a vu le jour il y a à peine deux décennies à Guelph, en Ontario, grâce au professeur Paul Hebert, pionnier et fondateur de l’initiative du Code-barres du vivant (Barcode of Life). L’idée lui est venue en observant les codes-barres sur les produits alimentaires : tout comme ces lignes verticales permettent de distinguer le lait entier du lait à 2 % à la caisse, il a imaginé un outil basé sur l’ADN capable d’identifier n’importe quelle espèce vivante sur Terre. Pendant des siècles — et encore aujourd’hui lorsque l’ADN ne peut être lu directement — l’identification des espèces repose sur l’examen de leurs caractéristiques anatomiques. Or, ces traits peuvent varier considérablement et exigent une expertise spécialisée pour être correctement interprétés.
L’initiative du Code-barres du vivant a marqué le début d’une véritable renaissance taxonomique à l’échelle mondiale. Des laboratoires aux quatre coins du globe se sont rapidement mobilisés pour séquencer le gène de la cytochrome oxydase I (COI) — la région d’ADN désignée comme norme internationale par Paul Hebert et ses collègues — dans le but de créer des bases de données de référence couvrant le plus grand nombre d’espèces possible. En un laps de temps étonnamment court, d’immenses bibliothèques de séquences COI ont été générées, regroupées et archivées dans des dépôts publics, accessibles à tous et soutenus par des financements gouvernementaux.
Malheureusement, des failles ont commencé à apparaître dans ce système. Dans la course décentralisée visant à séquencer et partager les gènes COI de toutes les espèces de la planète, des liens essentiels entre les séquences génétiques et les spécimens d’origine ont été perdus. Même lorsque ces liens ont été conservés, les précieux spécimens — qui permettent de relier les séquences COI à la taxonomie traditionnelle par l’analyse de leurs caractéristiques anatomiques — sont souvent conservés dans de petits laboratoires, éloignés ou sous-financés. Ces établissements ne peuvent pas toujours rendre les spécimens accessibles aux spécialistes en taxonomie, qui eux-mêmes manquent de ressources pour se déplacer et vérifier les identifications. En conséquence, les bibliothèques de référence de codes-barres existantes contiennent de nombreuses erreurs d’identification, et leur correction à grande échelle demeure extrêmement difficile, voire irréalisable.
Aujourd’hui, l’émergence du métabarcoding de l’ADNe marque le début d’une seconde révolution dans le domaine de la taxonomie moléculaire, avec des retombées majeures. Cette avancée ouvre de nouvelles perspectives, notamment pour la détection d’organismes rares, menacés ou potentiellement envahissants. Grâce aux technologies de séquençage génétique de nouvelle génération, les scientifiques spécialisés en biodiversité peuvent désormais identifier avec précision des centaines d’espèces simultanément, à partir de simples échantillons environnementaux — quelques litres d’eau ou même d’air. Ces échantillons contiennent des traces d’ADN issues des cellules mortes que tous les organismes multicellulaires libèrent continuellement dans leur environnement.
Travailler avec l’ADN environnemental, c’est un peu comme manipuler de la magie. On a l’impression d’avoir été propulsés dans un univers futuriste digne de Star Trek, où tout organisme ou objet peut être identifié instantanément grâce à un simple balayage de tricordeur. Bien que nous ne soyons encore loin d’effectuer des analyses d’ADN environnemental en temps réel, notre capacité actuelle à identifier, en quelques jours, plusieurs centaines d’espèces à partir d’un simple échantillon d’eau représente une avancée spectaculaire. Ce qui nécessitait auparavant des semaines, voire des mois de travail intensif sur le terrain peut désormais être accompli avec une rapidité et une précision inédites.
De manière fortuite, l’essor du métabarcoding offre aux scientifiques de la biodiversité une opportunité précieuse : celle de d’établir de nouvelles bases de données de référence en codes-barres, fondées sur des spécimens archivés, tout en corrigeant les lacunes de l’approche fragmentée qui prévalait jusqu’ici. En raison des limites techniques des méthodes de séquençage utilisées pour l’ADN environnemental, le gène COI — initialement proposé par Paul Hebert comme norme — a été remplacé par un gène voisin, le 12S. Cette transition nécessite la création de bases de données entièrement nouvelles pour cette région du code-barres, ouvrant ainsi une fenêtre stratégique que le ROM est particulièrement bien placé pour exploiter, grâce à sa vaste collection de spécimens entiers et d’échantillons génétiques congelés soigneusement documentés.
Chercheurs dans le domaine
Reconnaître ce besoin
Avant le lancement de notre projet, aucune séquence COI de poisson provenant de l’Amazonie équatorienne n’était disponible dans un vaste dépôt accessible au public, et seulement une séquence 12S. Depuis, notre initiative a permis de générer plus de 400 séquences COI et plus de 800 séquences 12S, et cette bibliothèque continue de s’enrichir.
Notre objectif est de créer une bibliothèque de référence combinée, intégrant les séquences 12S et COI, accessible au public, dans laquelle chaque séquence d’ADN est rigoureusement associée à un spécimen archivé et durablement conservé au ROM. Ainsi, lorsque cette bibliothèque sera utilisée pour identifier des échantillons d’ADN environnemental, les futures générations de scientifiques pourront retracer précisément l’origine de chaque séquence jusqu’au spécimen individuel correspondant. De plus, les chercheurs pourront également consulter ces spécimens sur place au ROM ou d’en demander le prêt pour approfondir leurs analyses. Cela permettra de réviser et d’actualiser les identifications taxonomiques au fil du temps, à mesure que notre compréhension de la biodiversité amazonienne et de la nomenclature évolue.
Lors d’un essai récent utilisant une première version de notre nouvelle bibliothèque de référence pour analyser des séquences d’ADN environnemental provenant de l’Amazonie équatorienne, les résultats ont été prometteurs. Nos données ont permis d’améliorer de plus de 60 % le taux d’identification précise des espèces, comparativement aux bases de données existantes qui ne comprenaient pas de références équatoriennes. Cependant, le nombre total d’espèces identifiées reste relativement faible : passant de 87 à 141, alors que les échantillons d’ADN environnemental indiquent la présence d’environ 410 espèces de poissons dans la région.
Même si nous réalisons des avancées considérables dans le développement de nouvelles méthodes d’étude de la biodiversité, la mise en place de l’infrastructure nécessaire pour exploiter pleinement ces outils demeure un défi de taille. Les années de travail, sur le terrain et dans les musées, nécessaires pour collecter, identifier et documenter chaque espèce afin d’établir une bibliothèque complète de spécimens et de séquences génétiques représentent une tâche monumentale. Pourtant, ce travail est fondamental. À une époque où l’espoir de préserver la nature peut sembler fragile, je suis plus enthousiaste que jamais à l’idée que les vastes collections d’histoire naturelle du ROM, accumulées sur plus d’un siècle, puissent être mobilisées pour répondre aux enjeux urgents posés par les approches modernes de classification et de conservation de nos voisins non humains.
Dans ce contexte, le terme « méta » désigne la capacité à lire simultanément des centaines de séquences d’ADN à partir d’un seul échantillon environnemental, une étape qui nécessite l’intervention d’un ordinateur pour distinguer et analyser chaque séquence individuelle.
Nathan K. Lujan
Nathan K. Lujan est conservateur associé des poissons au ROM. Le poste de conservateur de Nathan Lujan a été créé grâce au généreux soutien de la Herbert A. Fritch Family Foundation.